R ist sehr mächtig! Als Neuling, gezwungen durch die Arbeit oder aus Eigeninteresse, kann R auf den ersten Blick sehr komplex erscheinen. Es ist noch kein Meister vom Himmel gefallen und das gilt auch hier. In diesem Blog zeig ich eine einfache Datenaufbereitung.
Daseinsberechtigung R
Da du hier noch am Lesen bist hat du bereits schonmal von R gehört. R ist eine Open Source Programmiersprache für statistische Berechnung oder auch deren visuelle Darstellung und inzwischen eine feste Grösse. Im Zusammenhang mit Big Data Analytics ist R kaum wegzudenken. R ist die Programmiersprache und R Studio ist die grafische Benutzeroberfläche dazu.
Suchmaschinen sind dein neuer bester Freund
Das Rad muss nicht neu erfunden werden und somit auch der R Code nicht. Viele Fragen klären sich indem sich zeitgenommen wird und via Suchmaschinen die entsprechenden Codeteile gesucht respektive auch übernommen werden.
Daten
Bevor wir starten können brauchen wir einen Datensatz den wir aufbereiten wollen. Damit du dies nachvollziehen kannst nehme ich einen Datensatz von Opendata.swiss. Für das Beispiel verwenden wir Impfdaten. In diesem Datensatz sind die Anzahl der Geimpften nach Impfstoff, Impfstatus, etc. aufgeteilt. Für jeden Tag gibt es mehrere Einträge pro Impfstoff. Da ich kein Fan von Pivo Tabellen bin, erstelle ich nun eine Tabelle (Data Frame) mit nur noch einem Eintrag pro Tag.
Die erste zwei Zeilen Code
In RStudio eröffnen wir als erstes ein neues File (File/New File/R Script). Nun können wir loslegen! Um das File laden zu können setzten wir das Working Directory (setwd) mit folgendem Code:
setwd(„C:/Users/Beispiel“)
Diese Zeile kann nun markiert und via CTR + Enter (auf Windows) ausgeführt werden.
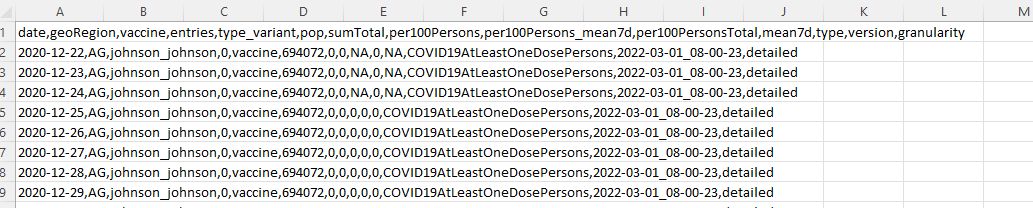
Die zweite Zeile ist genau so unspektakulär wie die erste. Mit dem folgenden Code lesen wir das File (read.table) und weisen dies einem Namen (Variablen) zu (<-).
impfungen <- read.table(„COVID19VaccPersons_vaccine.csv“, header = TRUE, sep = „,“)
Im Code haben wir noch als Parameter den Separater, Komma, sowie mitgegeben, dass die erste Zeile als Header gelesen werden soll. Nun habe ich das .csv File in zwei einfach schritten ins RStudio geladen.
Filtern wie im Excel
Als nächstes interessiert uns nur die Region CH (filter()). Im Filter geben wir die entsprechen Spalte an die wir filtern möchten (geoRegion==“CH“) und überschreiben den Datensatz mit den gefilterten Daten.
impfungen <- filter(impfungen, geoRegion==“CH“)
Nun scheint es “komplizierter” zu werden
Als nächsten erstellen wir die neu angeordnete Tabelle (data frame) in einem Schritt. Als erstes legen wir die erst Spalte fest indem wir dieser das Datum zuweisen. Mittels «Subset» weisen wir die täglichen Impfungen von Pfizer zu. Die spalten werden via $ eines Datensatz ausgelesen. Den gleichen Vorgang machen wir für alle Impfstoffe. Natürlich sind hier die Möglichkeiten gegen oben offen.
impfungen <- data.frame(
impfungen$date,
subset(impfungen$entries,impfungen$vaccine == „pfizer_biontech“ & impfungen$type ==“COVID19FullyVaccPersons“),
subset(impfungen$entries,impfungen$vaccine == „johnson_johnson“ & impfungen$type ==“COVID19FullyVaccPersons“),
subset(impfungen$entries,impfungen$vaccine == „moderna“ & impfungen$type ==“COVID19FullyVaccPersons“)
)
Und schon fast fertig
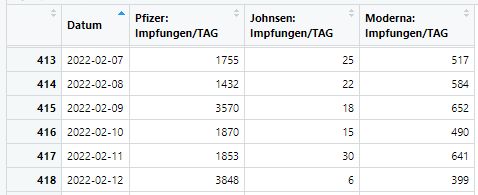
Zum Schluss weisen wir den Spalten noch neue Namen (colnames) zu. Zusätzlich werden doppelte Einträge (distinct) aus dem Datensatz entfernt. Die letzte Zeile Code spricht nun für sich.
colnames(impfungen)[1] <- „Datum“
colnames(impfungen)[2] <- „Pfizer: Impfungen/TAG“
colnames(impfungen)[3] <- „Johnsen: Impfungen/TAG“
colnames(impfungen)[4] <- „Moderna: Impfungen/TAG“
impfungen<-distinct(impfungen)
write.csv(impfungen,(„C:/Users/Beispiel/impfungen.csv“)
Fazit
Mit der richtigen Anleitung können einfache Sachen relativ schnell umgesetzt werden. Dies weil die Community relativ gross ist und mit der richtigen Frage in einer Suchmaschine der entrechtende Code schnell gefunden wird. Es wird jedoch schnell komplex. So empfiehlt es sich genug Zeit einzuplanen, wenn mit R gearbeitet werden möchte. Um Codefragmente besser zu verstehen lohnt es sich auch Video Turtorials zu schauen, da bei solchen Videos der Code teileweise sehr gut erklärt wird.
